BSTEM
PROGRAMMA DIAGNOSTICO, STATISTICO E DIDATTICO
PER IL TRONCO CEREBRALE
Il “tronco cerebrale” o “troncoencefalo”, porzione di sistema nervoso interposta tra midollo spinale ed encefalo all’altezza della sommità del collo, è una piccola struttura che svolge centinaia di funzioni. Qui, in gruppi stretti gli uni agli altri, giacciono i nuclei e le radici dei nervi cranici, e le vie lunghe, sensoriali e motorie, che interconnettono il cervello, il cervelletto, e il midollo spinale.
Lesioni focali nel troncoencefalo danno origine a insiemi di sintomi e segni tali che nella pratica clinica localizzare una lesione troncoencefalica è spesso un compito complesso anche per un neurologo esperto. Le immagini neuroradiologiche (RM oTAC) non sempre aiutano, poiché presentano una bassa correlazione con i sintomi e segni clinici in particolare nelle lesioni demielinizzanti e infiammatorie, tanto che si parla sovente di “lesione funzionale” distinta dalla “lesione anatomica”. Indagini neurofisiologiche (potenziali evocati e riflessi trigeminali) aggiungono importanti informazioni topodiagnostiche, ma, senza un modo per mappare la lesione funzionale, anche l’interpretazione dei risultati neurofisiologici rimane un difficile compito lasciato al giudizio soggettivo di un neurofisiologo esperto con speciale conoscenza del troncoencefalo.
Tradurre, in modo oggettivo e riproducibile, i dati clinici e i risultati neurofisiologici in una immagine della corrispondente lesione funzionale, porta almeno due notevoli benefici: rende più agevolmente comprensibili i risultati dei tests neurofisiologici, favorendone così l’utilizzo e la diffusione; e rende possibile confrontare quantitativamente/statisticamente la lesione funzionale e la lesione anatomica, così da poter studiare la loro correlazione anche in rapporto al tipo di patologia.
BSTEM è un programma diagnostico, didattico e di ricerca capace appunto di tradurre i dati clinici e neurofisiologici del troncoencefalo in immagini 2D e 3D di lesione, e di analizzare statisticamente lesioni troncoencefaliche anatomiche o neuroradiologiche per studiarne la corrispondenza con compromissioni funzionali (alterazioni dei test neurofisiologici), in modo da ottenere informazioni sulla collocazione topografica di quelle funzioni.
Uso diagnostico e didattico
In questo uso il programma viene utilizzato per fornire immagini di lesione a partire dai dati clinici e neurofisiologici.
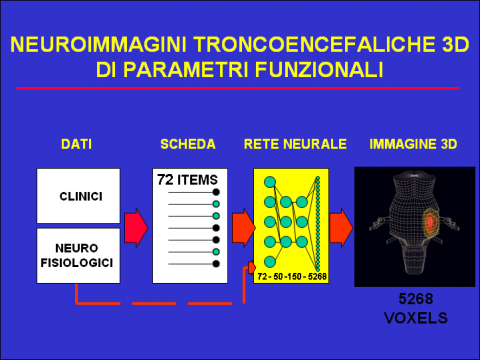
I dati clinici (sintomi e segni rilevati all’esame neurologico) e neurofisiologici (riflessi trigeminali e potenziali evocati acustici, motori e somatosensoriali) vengono forniti dall’utente al programma tramite una scheda di 72 item (23 dati clinici e 13 dati neurofisiologici per ciascuno dei due lati del corpo), a ognuno dei quali l’utente assegna un valore normale, dubbio, patologico, o non esaminato.
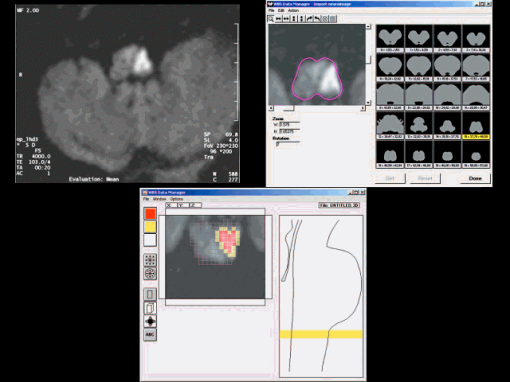
Da questi dati il programma ricostruisce una lesione mappandola su un modello di troncoencefalo ideale suddiviso in 5268 microvolumi elementari (“voxels”) da 2x2x2 mm a 2x2x4 mm. La mappatura consiste nell’assegnare a ciascun voxel un’appropriata probabilità, rappresentata in codice-colore, di essere coinvolto dalla lesione. Ne risulta una ricostruzione tridimensionale, in falsi colori, della lesione probabile, che il programma può poi ulteriormente elaborare matematicamente (filtraggi, windowing, arrotondamenti, ecc.), ruotare, mostrare da ogni punto di vista, o esportare verso programmi di computer-grafica come 3D-Studio a fini didascalici. Naturalmente, possono anche essere estratte sezioni bidimensionali assiali, sagittali e coronali da esaminare come le comuni sezioni nuroradiologiche.
RETE NEURALE
La diagnosi viene fornita da una rete neurale multistrato, feed-forward, completamente connessa, con due strati nascosti, addestrata tramite back-propagation.
Tale rete ha 144 unità di input, corrispondenti ai 72 item clinici e neurofisiologici, ognuno codificato su due unità binarie nei suoi 4 possibili valori (normale, dubbio, patologico, o non esaminato). Ha poi 50 unità nel primo strato nascosto, 150 unità nel secondo, e 2634 unità di output.
Queste 2634 unità di output corrispondono al mezzo troncoencefalo di un solo lato: grazie alla simmetria destra-sinistra del troncoencefalo e a un processo di attivazione della rete in due passi, abbiamo potuto dimezzare le 5268 unità di output richieste dal modello 3D di troncoencefalo completo. In un primo passo la rete calcola i valori di output per i voxel dell’emitronco sinistro; in un secondo passo essa viene attivata con valori di input invertiti di lato, e i valori di output vengono assegnati ai voxel dell’emitronco destro.
La rete è stata addestrata con 580 casi-esempio rappresentativi delle principali possibili combinazioni di dati clinici e neurofisiologici, e delle più tipiche lesioni troncoencefaliche. Ogni caso aveva una sola lesione focale. A ciascun voxel di ogni caso-esempio è stata assegnata una probabilità locale di lesione su una scala di tre valori: 0 = sano; 0.5 = dubbio; 1 = colpito. Altri 200 casi-esempio, allestiti nello stesso modo, sono stati riservati come casi-test. Tutti questi casi sono stati selezionati da tre esperti di anatomia e fisiologia del troncoencefalo come presentanti buona compatibilità tra la lesione e i dati clinici e neurofisiologici.
L’apprendimento, attuato con velocità di apprendimento variabile in funzione dell’entità complessiva di variazione dei pesi, è stato arrestato quando il valore fornito dalla rete in ogni voxel è arrivato entro 0.25 dal valore desiderato (circa 1 mese su un computer Pentium II a 350 MHz).
Al termine dell’apprendimento la rete è stata valutata sui 200 casi riservati come test. La differenza tra output della rete e valore desiderato è risultata inferiore a 0.25 – cioè tale da poter classificare correttamente un voxel come sano o dubbio o leso – in oltre l’83% dei voxel; percentuale che sale al 96% se “si perdonano” differenze spaziali entro un voxel di distanza, e al 99.6% entro due voxel di distanza (cioè molti errori sono in realtà piccoli errori spaziali).
Ad apprendimento ultimato un esperto ha anche valutato le diagnosi della rete su altri 37 pazienti con lesione troncoencefalica singola non tumorale e non demielinizzante (patologie note per presentare scarsa correlazione anatomo-funzionale), riscontrando la diagnosi corretta in 31 casi. I restanti 6 pazienti presentavano solo segni clinici o neurofisiologici di interessamento delle vie lunghe motorie o sensoriali di un solo lato (emisindrome piramidale o somatosensoriale), e la rete ha assegnato valori uniformi di probabilità di lesione a tutti i voxel del tratto corticospinale o somatosensoriale coinvolto. Questo non è un errore, ma l’ovvia conseguenza della mancanza di un segno “di livello” capace di consentire una più precisa localizzazione longitudinale della lesione.
Uso di ricerca
In questo uso il programma viene utilizzato per valutare statisticamente l’associazione tra la compromissione di un test neurofisiologico e la collocazione di una lesione troncoencefalica neuroradiologicamente documentata, al fine di identificare con precisione quali aree troncoencefaliche sono responsabili per quel segno neurofisiologico.
Qui l’utente non fornisce al programma i dati clinici e neurofisiologici, ma invece introduce nel modello ideale 3D di troncoencefalo il valore di lesione di ciascun voxel (0 = voxel sano; 0.5 = voxel dubbio; 1 = voxel leso) di un gruppo di pazienti con un dato indice neurofisiologico compromesso e con lesione troncoencefalica neuroradiologicamente documentata. Il programma si incarica di interpolare ed estrapolare i dati neuroradiologici 2D inseriti trasformandoli in dati 3D.
Il valore medio di lesione di ciascun voxel così ottenuto (somma del valore nei singoli pazienti, divisa per il numero di pazienti) viene confrontato statisticamente con il valore medio di quel medesimo voxel in un gruppo di controllo (test a due campioni), oppure con la media di tutti i voxel di tutti i pazienti del gruppo patologico, media che rappresenta la probabilità generica di un voxel qualsiasi di essere sede di lesione nel gruppo patologico (test a un campione). In ambedue i casi l’immagine di lesione restituita dal programma, ancora distribuita in codice-colore nel modello ideale 3D di troncoencefalo, rappresenta la distribuzione topografica delle significatività statistiche riscontrate, consentendo così di identificare i punti di massima associazione statistica tra lesione neuroradiologica e segno funzionale. Quelli saranno i punti maggiormente responsabili ai fini della funzione neurofisiologica studiata.
Se l’importazione dei dati neuroradiologici nel troncoencefalo ideale è fatta con cura e i test statistici sono applicati correttamente, questo è un metodo di studio molto potente e di grande sensibilità. Il programma consente quindi di deformare il troncoencefalo ideale per adattarlo con precisione alle immagini neuroradiologiche del singolo paziente nella fase di immissione dei dati, e alle immagini di atlanti di riferimento, oltre che ovviamente ancora del singolo paziente, nella fase di emissione dei risultati; e adotta test statistici non parametrici (Chi-quadro e Kolmogorov-Smirnov negli studi a un campione, Chi-quadro e test U di Mann-Whitney negli studi a due campioni) idonei a questo tipo di studio.
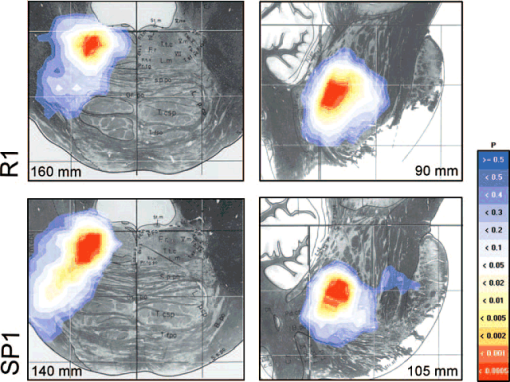
Le seguenti figure illustrano a titolo esemplificativo i risultati di uno studio dei riflessi R1, R2, SP1, SP2 e JJ (riflessi rispettivamente precoce e tardivo di ammiccamento, precoce e tardivo di inibizione del massetere, e “jaw jerk”) in un lavoro di Cruccu et al. (“Brainstem reflex circuits revisited”, Brain, 2005;128:386-94).
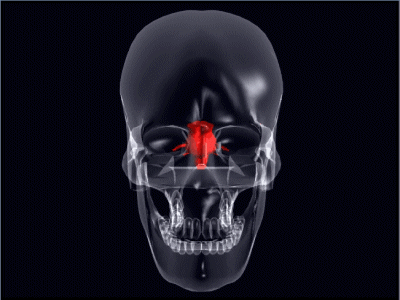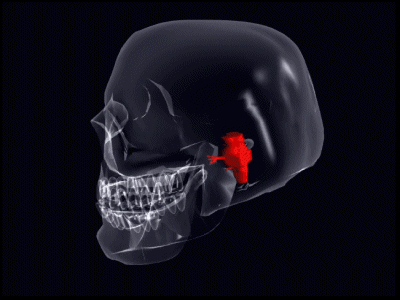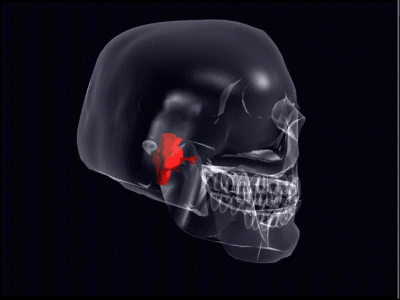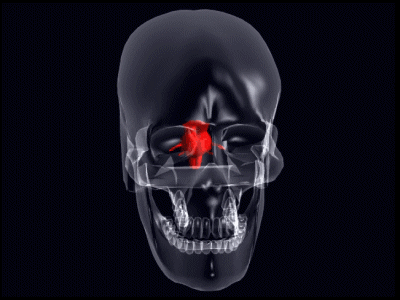
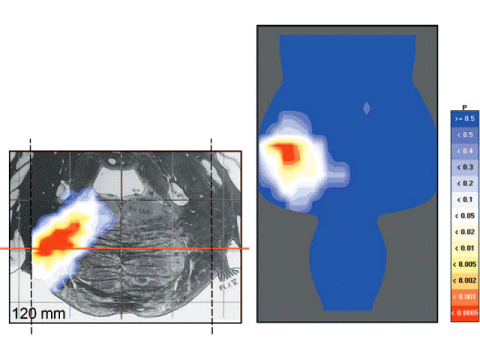
Riflessi R1 e SP1 in sezione pontina assiale (a sinistra) e sagittale (a destra). Ambedue questi riflessi risultano avere i loro voxel più significativi nel ponte intermedio dorsale, vicino al pavimento del IV ventricolo e al nucleo sensitivo trigeminale principale, con scarse differenze tra i due. Sebbene i voxel più significativi di R1 appaiano lievemente più mediali e caudali di quelli di SP1, una successiva verifica su 13 pazienti con R1 alterato e SP1 normale, e 23 pazienti con R1 normale e SP1 alterato, ha rivelato che tale differenza non è significativa.
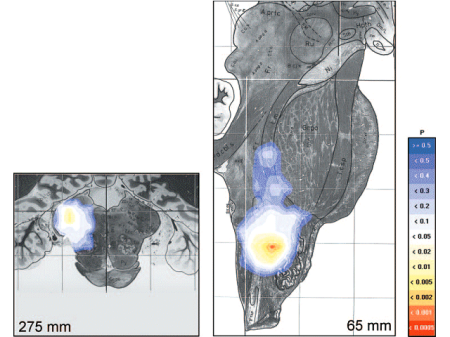
Riflesso R2 afferente in sezione assiale (a sinistra) e sagittale (a destra). Il cuore della significatività risulta nel midollo laterale a livello del nucleo ambiguo, dell’emergenza delle radici del IX e X nervo cranico, e dell’oliva inferiore, senza scendere sotto il polo caudale del nucleo olivare inferiore.
I riflessi precoci (R1, SP1 e JJ) in sezione assiale (a sinistra) e coronale (a destra). L’area significativa disegna il decorso intrapontino delle vie afferenti primarie trigeminali.